So far, we have looked at the mechanism by which the information in genes (DNA) is transcribed into RNA. O RNA recém-feito, também conhecido como a transcrição primária (o produto da transcrição é conhecido como uma transcrição) é processado posteriormente antes de ser funcional. Tanto os procariontes quanto os eucariontes processam seus ribossômicos e transferem RNAs.
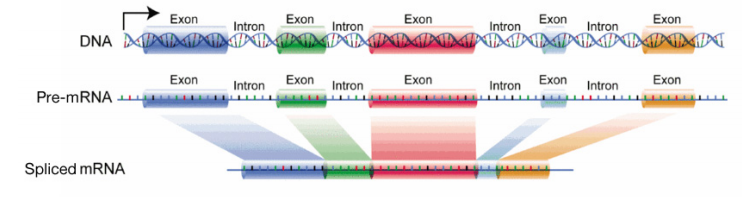
a principal diferença no processamento de RNA, no entanto, entre procariontes e eucariontes, está no processamento de RNAs mensageiro. Vamos concentrar-nos no processamento do mRNAs nesta discussão. Deve lembrar-se que, nas células bacterianas, o ARNm é traduzido directamente à medida que sai do modelo de ADN. Nas células eucarióticas, a síntese de RNA, que ocorre no núcleo, é separada da máquina de síntese de proteínas, que está no citoplasma. Além disso, os genes eucarióticos têm intrões, regiões não codificadas que interrompem a sequência de codificação do gene., O ARNm copiado de genes que contêm intrões também terá, portanto, regiões que interrompem a informação no gene. Estas regiões devem ser removidas antes de o ARNm ser enviado para fora do núcleo para ser usado para dirigir a síntese de proteínas. O processo de remoção dos introns e de junção das seções de codificação ou exons, do RNAm, é chamado splicing. Uma vez que o ARNm foi tapado, esplicado e teve uma cauda polyA adicionada, ele é enviado do núcleo para o citoplasma para tradução.
O produto inicial da transcrição de um gene que codifica proteínas é chamado de pré-mRNA (ou transcrição primária)., Depois de ter sido processado e está pronto para ser exportado do núcleo, é chamado de mRNA Maduro ou mRNA processado.
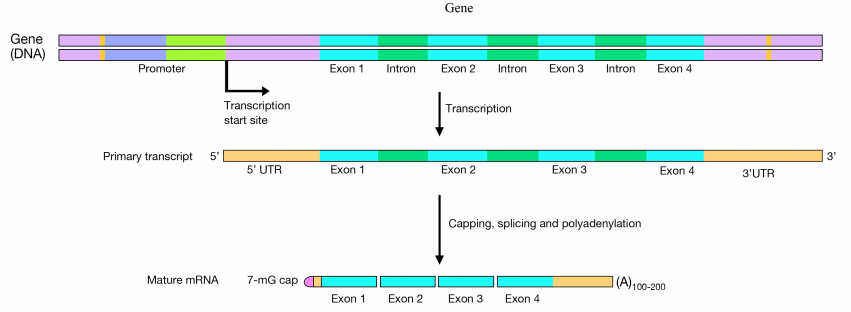
o Que são os passos do processamento de Rna mensageiro?nas células eucarióticas, as pré-mRNAs passam por três fases principais de processamento:
- tapping at the 5′ end
- adição de uma cauda de polyA no 3′ end., na fase de nivelamento do processamento de mRNA, adiciona-se uma guanosina de 7-metila (mostrada à esquerda) na extremidade de 5′ do mRNA. A tampa protege a extremidade 5′ do ARNm da degradação pelas nucleases e também ajuda a posicionar o ARNm corretamente nos ribossomas durante a síntese de proteínas.
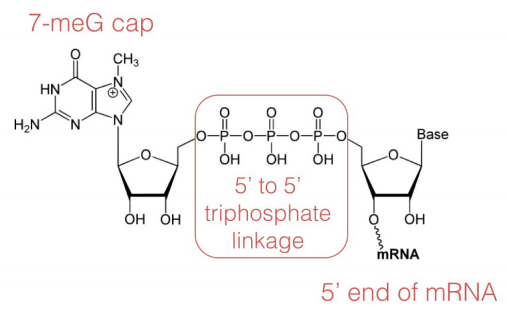
Figura 5.5.,3: mRNA capping structure The 3′ end of a eukaryotic mRNA is first trimmed, then an enzyme called PolyA Polymerase adds a “tail” of about 200 ‘a’ nucleotides to the 3′ end. Há evidências de que a cauda polyA desempenha um papel na tradução eficiente do ARNm, bem como na estabilidade do ARNm. A tampa e a cauda de polyA em um ARNm também são indicações de que o ARNm está completo (isto é, não defeituoso). Os intrões são removidos do pré-ARNm pela atividade de um complexo chamado spliceosome., O spliceossoma é composto por proteínas e pequenas RNAs que estão associadas a formar enzimas proteínas-ARN chamadas pequenas ribonucleoproteínas nucleares ou snRNPs (SNURPS pronunciadas). A máquina de costura deve ser capaz de reconhecer as junções de junção (isto é, o fim de cada exon e o início do próximo), a fim de cortar corretamente os intrões e juntar os exons para fazer o mRNA Maduro, spliced.que sinais indicam onde um intron começa e termina? A sequência de base no início (5′ ou extremidade esquerda, também chamado de local doador) de um intrão é GU, enquanto a sequência no final 3′ ou direita (a. k. a.,
= = ligações externas = = Há também uma terceira sequência importante dentro do intron, chamada de ponto de ramificação, que é importante para o splicing.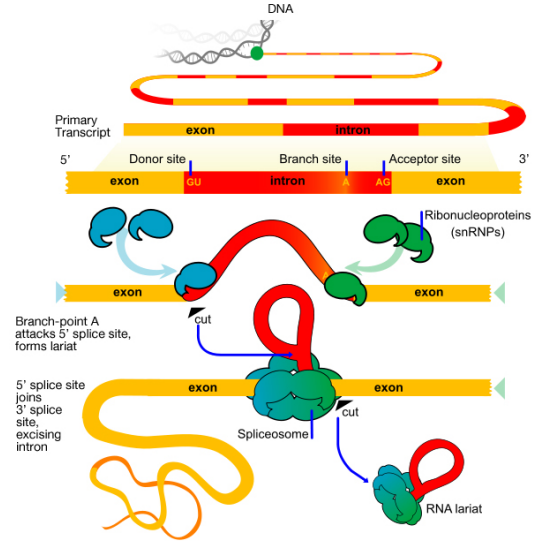
Figura 5.5.4: Splicing Há duas etapas principais no processo de splicing:
- Na primeira etapa, o pré-mRNA é cortado em 5′ splice site (junção dos 5′ exão e o intron). O fim de 5′ do intron então é unido ao ponto do ramo dentro do intron., Isto gera a molécula em forma de lariat característica do processo de splicing
- na segunda etapa, o local de splice 3′ é cortado, e os dois exons são unidos, e o intron é liberado.
muitos pré-mRNAs têm um grande número de exons que podem ser agrupados em diferentes combinações para gerar diferentes mRNAs maduros. Isto é chamado de splicing alternativo, e permite a produção de muitas proteínas diferentes usando relativamente poucos genes, uma vez que um único RNA pode, combinando diferentes exons durante splicing, criar muitas mensagens de codificação de proteínas diferentes., Por causa de splicing alternativo, cada gene em nosso DNA dá origem, em média, a três proteínas diferentes. Uma vez que as mensagens de codificação de proteínas tenham sido processadas por tapping, splicing e adição de uma cauda poli a, elas são transportadas para fora do núcleo para serem traduzidas no citoplasma.
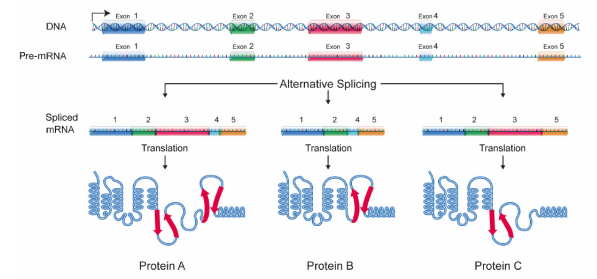
Figura 5.5.5: Splicing e a diversidade proteica Contribuintes
-
o Dr. Kevin Ahern e Dr. Indira Rajagopal (Universidade do Estado de Oregon)