Finora, abbiamo esaminato il meccanismo con cui le informazioni nei geni (DNA) vengono trascritte in RNA. L’RNA appena fatto, noto anche come trascrizione primaria (il prodotto della trascrizione è noto come trascrizione) viene ulteriormente elaborato prima che sia funzionale. Sia i procarioti che gli eucarioti elaborano i loro RNA ribosomiali e trasferiscono.
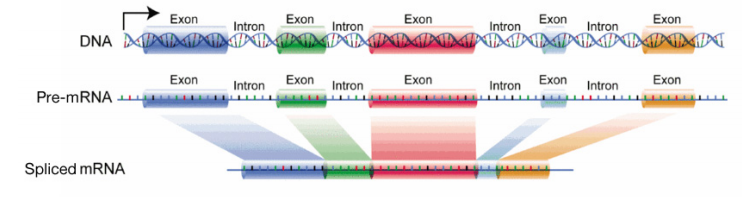
La principale differenza nell’elaborazione dell’RNA, tuttavia, tra procarioti ed eucarioti, è nell’elaborazione degli RNA messaggeri. In questa discussione ci concentreremo sull’elaborazione degli MRNA. Ricorderai che nelle cellule batteriche, l’mRNA viene tradotto direttamente mentre si stacca dal modello del DNA. Nelle cellule eucariotiche, la sintesi dell’RNA, che si verifica nel nucleo, è separata dal macchinario di sintesi proteica, che si trova nel citoplasma. Inoltre, i geni eucariotici hanno introni, regioni non codificanti che interrompono la sequenza codificante del gene., L’mRNA copiato da geni contenenti introni avrà anche regioni che interrompono le informazioni nel gene. Queste regioni devono essere rimosse prima che l’mRNA venga inviato fuori dal nucleo per essere utilizzato per dirigere la sintesi proteica. Il processo di rimozione degli introni e ricongiungimento delle sezioni di codifica o esoni, dell’mRNA, è chiamato splicing. Una volta che l’mRNA è stato tappato, impiombato e aveva una coda polyA aggiunto, viene inviato dal nucleo nel citoplasma per la traduzione.
Il prodotto iniziale della trascrizione di un gene codificante per proteine è chiamato pre-mRNA (o trascrizione primaria)., Dopo che è stato elaborato ed è pronto per essere esportato dal nucleo, è chiamato mRNA maturo o mRNA elaborato.
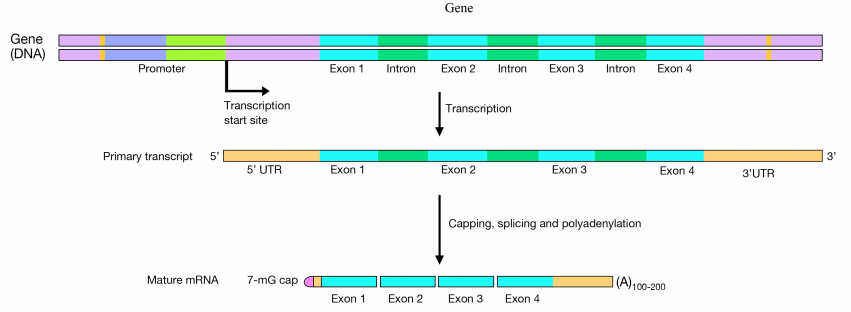
Quali sono le fasi di elaborazione per messenger RNA?
Nelle cellule eucariotiche, i pre-mRNA subiscono tre fasi principali di lavorazione:
- Capping all’estremità 5′
- Aggiunta di una coda di polyA all’estremità 3′., e
- Splicing per rimuovere gli introni
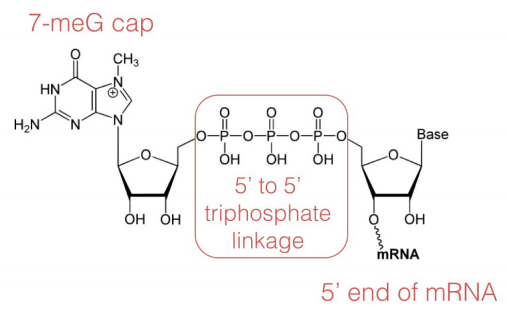
Nella fase di tappatura dell’elaborazione dell’mRNA, una 7-metil guanosina (mostrata a sinistra) viene aggiunta all’estremità 5′ dell’mRNA. Il cappuccio protegge l’estremità 5′ dell’mRNA dalla degradazione dalle nucleasi e aiuta anche a posizionare correttamente l’mRNA sui ribosomi durante la sintesi proteica.
L’estremità 3′ di un mRNA eucariotico viene prima tagliata, quindi un enzima chiamato POLYA Polimerasi aggiunge una” coda “di circa 200 nucleotidi ‘A’ all’estremità 3′. Ci sono prove che la coda polyA svolge un ruolo nella traduzione efficiente dell’mRNA, così come nella stabilità dell’mRNA. Il cappuccio e la coda di polyA su un mRNA sono anche indicazioni che l’mRNA è completo (cioè non difettoso). Gli introni vengono rimossi dal pre-mRNA dall’attività di un complesso chiamato spliceosoma., Lo spliceosoma è costituito da proteine e piccoli RNA che sono associati a formare enzimi proteici-RNA chiamati piccole ribonucleoproteine nucleari o snRNPs (SNURPS pronunciati). La macchina di giunzione deve essere in grado di riconoscere le giunzioni di giunzione (cioè la fine di ciascun esone e l’inizio del successivo) al fine di tagliare correttamente gli introni e unire gli esoni per ottenere l’mRNA maturo e impiombato.
Quali segnali indicano dove inizia e finisce un introne? La sequenza di base all’inizio (5′ o estremità sinistra, chiamata anche sito donatore) di un introne è GU mentre la sequenza all’estremità 3 ‘ o destra (aka., sito accettore) è AG. C’è anche una terza sequenza importante all’interno dell’introne, chiamata punto di diramazione, che è importante per lo splicing.
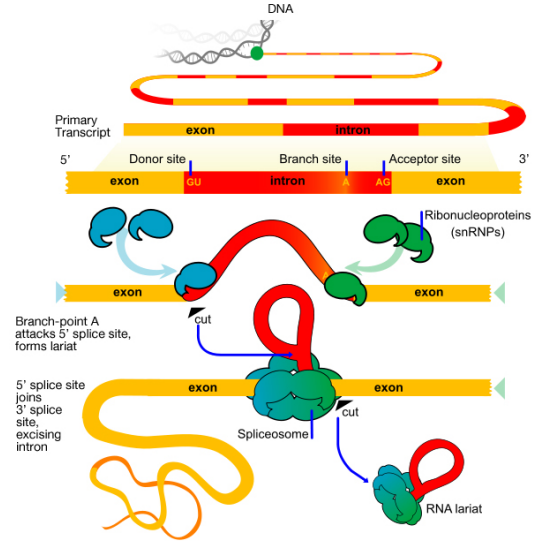
Ci sono due passaggi principali nello splicing:
- Nel primo passaggio, il pre-mRNA viene tagliato a il sito di giunzione 5′ (la giunzione dell’esone 5′ e dell’introne). L’estremità 5 ‘ dell’introne viene quindi unita al punto di diramazione all’interno dell’introne., Ciò genera la caratteristica a forma di lariat della molecola del processo d’impionbatura
- Nel secondo punto, il sito della giuntura 3′ è tagliato ed i due esoni sono uniti insieme e l’introne è liberato.
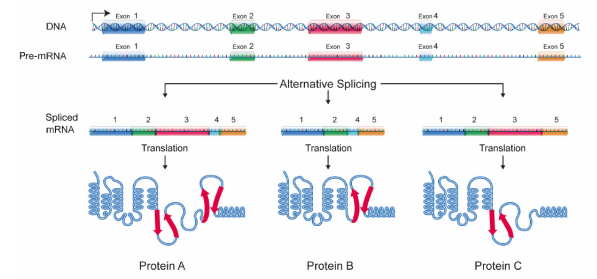
Molti pre-mRNA hanno un gran numero di esoni che possono essere uniti in diverse combinazioni per generare diversi MRNA maturi. Questo è chiamato splicing alternativo e consente la produzione di molte proteine diverse utilizzando relativamente pochi geni, poiché un singolo RNA può, combinando diversi esoni durante lo splicing, creare molti diversi messaggi di codifica proteica., A causa dello splicing alternativo, ogni gene nel nostro DNA dà origine, in media, a tre diverse proteine. Una volta che i messaggi di codifica delle proteine sono stati elaborati mediante capping, splicing e aggiunta di una coda poli A, vengono trasportati fuori dal nucleo per essere tradotti nel citoplasma.
Collaboratori
-
Dr. Kevin Ahern e Dr. Indira Rajagopal (Università di Stato dell’Oregon)