hasta ahora, hemos analizado el mecanismo por el cual la información en los genes (ADN) se transcribe en ARN. El ARN recién hecho, también conocido como transcripción primaria (el producto de la transcripción se conoce como transcripción) se procesa posteriormente antes de que sea funcional. Tanto procariotas como eucariotas procesan sus ARN ribosómicos y los transfieren.
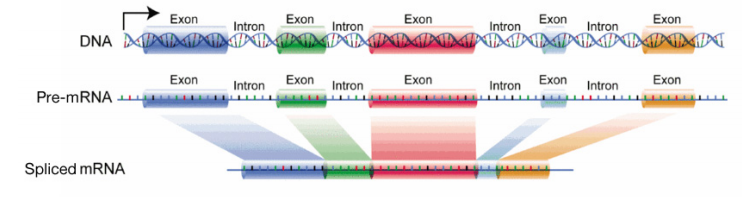
la principal diferencia en el procesamiento de ARN, sin embargo, entre procariotas y eucariotas, está en el procesamiento de ARN mensajeros. Nos centraremos en el procesamiento de los ARNm en esta discusión. Usted recordará que en las células bacterianas, el ARNm se traduce directamente a medida que sale de la plantilla de ADN. En las células eucariotas, la síntesis de ARN, que se produce en el núcleo, se separa de la maquinaria de síntesis de proteínas, que está en el citoplasma. Además, los genes eucariotas tienen intrones, regiones no codificantes que interrumpen la secuencia de codificación del gen., Por lo tanto, el ARNm copiado de genes que contienen intrones también tendrá regiones que interrumpen la información en el gen. Estas regiones deben eliminarse antes de que el ARNm se envíe fuera del núcleo para ser utilizado para dirigir la síntesis de proteínas. El proceso de eliminar los intrones y volver a unir las secciones de codificación o exones, del ARNm, se llama empalme. Una vez que el ARNm ha sido tapado, empalmado y se le ha añadido una cola de polyA, se envía desde el núcleo al citoplasma para su traducción.
el producto inicial de la transcripción de un gen codificador de proteínas se llama pre-ARNm (o transcripción primaria)., Después de que ha sido procesado y está listo para ser exportado desde el núcleo, se llama ARNm maduro o ARNm procesado.
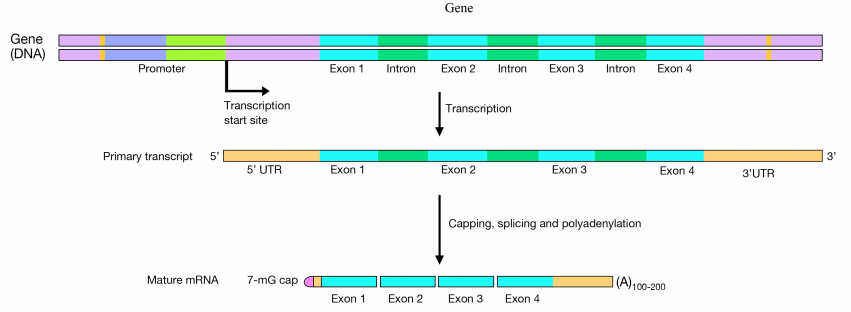
¿cuáles son los pasos de procesamiento de Arn mensajero?
en células eucariotas, los pre-ARNm se someten a tres pasos principales de procesamiento:
- tapado en el extremo 5′
- adición de una cola de polyA en el extremo 3′., y
- empalme para eliminar intrones
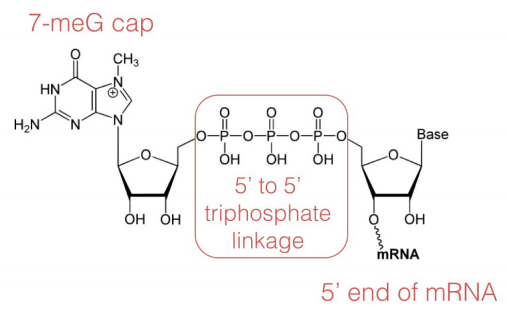
en la etapa de tapado del procesamiento de ARNm, se agrega una 7-metil guanosina (mostrada a la izquierda) en el extremo 5′ del ARNm. La tapa protege el extremo 5′ del ARNm de la degradación por nucleasas y también ayuda a posicionar el ARNm correctamente en los ribosomas durante la síntesis de proteínas.
el extremo 3′ de un ARNm eucariótico se recorta primero, luego una enzima llamada PolyA polimerasa agrega una» cola » de aproximadamente 200 nucleótidos ‘A’ al extremo 3′. Hay evidencia de que la cola de polyA juega un papel en la traducción eficiente del ARNm, así como en la estabilidad del ARNm. La tapa y la cola de polyA en un ARNm también son indicaciones de que el ARNm está completo (es decir, no defectuoso). Los intrones se eliminan del pre-ARNm por la actividad de un complejo llamado spliceosoma., El spliceosoma está compuesto por proteínas y pequeños ARN que se asocian para formar enzimas proteína-ARN llamadas pequeñas ribonucleoproteínas nucleares o snRNPs (pronunciadas SNURPS). La maquinaria de empalme debe ser capaz de reconocer las uniones de empalme (es decir, el final de cada exón y el comienzo del siguiente) para cortar correctamente los intrones y unir los exones para hacer el ARNm maduro y empalmado.
¿Qué señales indican dónde comienza y termina un intrón? La secuencia de base al principio (5′ o extremo izquierdo, también llamado el sitio donante) de un intrón es GU, mientras que la secuencia en el extremo 3′ o derecho (a. k.a., acceptor site) es AG. También hay una tercera secuencia importante dentro del intrón, llamada punto de ramificación, que es importante para el empalme.
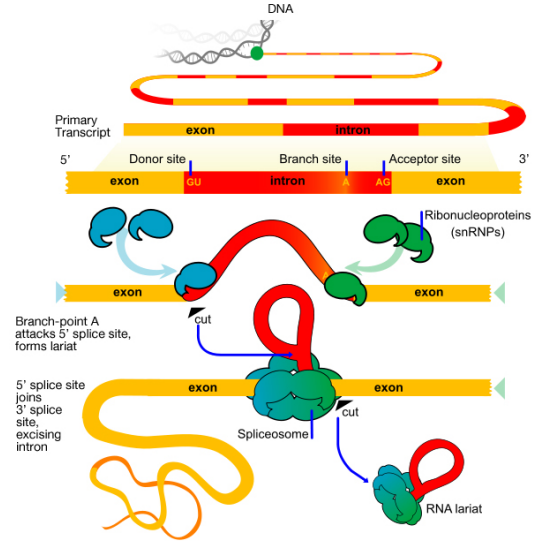
hay dos pasos principales en el empalme:
- En el primer paso, el pre-mRNA se corta en el sitio de empalme de 5′ (la unión del exón de 5′ y el intrón). El final de 5′ del intrón se une al punto de ramificación dentro del intrón., Esto genera la molécula en forma de lazo característica del proceso de empalme
- En el segundo paso, se corta el sitio de empalme de 3′, se unen los dos exones y se libera el intrón.
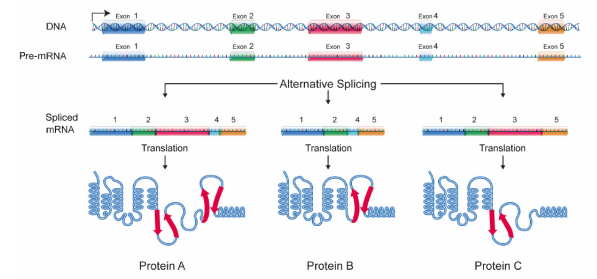
muchos pre-ARNm tienen un gran número de exones que se pueden empalmar juntos en diferentes combinaciones para generar diferentes ARNm Maduros. Esto se llama empalme alternativo, y permite la producción de muchas proteínas diferentes usando relativamente pocos genes, ya que un solo ARN puede, al combinar diferentes exones durante el empalme, crear muchos mensajes de codificación de proteínas diferentes., Debido al empalme alternativo, cada gen en nuestro ADN da lugar, en promedio, a tres proteínas diferentes. Una vez que los mensajes de codificación de proteínas han sido procesados por tapado, empalme y adición de una cola de poli A, son transportados fuera del núcleo para ser traducidos en el citoplasma.
colaboradores
-
Dr. Kevin Ahern y Dr. Indira Rajagopal (Oregon State University)